גני HOX
גֵני HOX הם קבוצה גדולה של גנים המקודדים לפקטורי שעתוק, המעורבים בהכוונת ההיווצרות של צירי הגוף ויצירת האיברים במהלך ההתפתחות העוברית של כל היצורים מרובי האיברים שנחקרו עד היום[1]. גנים אלה התגלו לראשונה במסגרת מחקר של פגמים התפתחותיים בתסיסנית המחקר (דרוזופילה מלנוגסטר). גני HOX התגלו ברוב האאוקריוטים שנבחנו אך לא בחיידקים וארכיאה[2]. כאשר רצף ה-DNA של גנים אלה פוענח, התברר כי כולם חולקים אזור מבני באורך של כ-180 בסיסים[3], המכונה homeodomain[4] (אנ'). אורך ה-homeodomain כ־60 חומצות אמינו, ותפקידו לאפשר לחלבון ה-HOX להיקשר לאתרים בקרתיים (אנ') ב-DNA .קישור זה מאפשר לחלבוני ה-HOX לתפקד כפקטורי שעתוק המבקרים שעתוק של גני המטרה שלהם, המשחקים תפקיד בבקרה של תהליכים התפתחותיים[5].
פעילות ביולוגית
עריכהגני HOX מהווים משפחה גדולה של גנים ששולטים בהתמיינות של תאים[6] ויצירת איברים במהלך התפתחות עוברית, ועל כן נחשבים ל "מבקרי-על"[7]. גני ה-HOX אחראיים ליצירת צירי הגוף הקדמיים והאחוריים של העובר, למיקום איברי הגוף בהתפתחות עוברית מוקדמת[8]. בצמחים, החלבונים המקודדים על ידי גני ה-HOX משחקים תפקיד חשוב בבקרה על תהליכים התפתחותיים, כגון תחזוקה של תאי גזע, התפתחות רוחבית, תגובה לעקות ותגובה לאור[9][10][11].
יש דמיון בתהליכים הגנטיים המבקרים התפתחות עוברית ותהליך יצירת גידול סרטני, ולכן נתגלתה מעורבות של פעילות לא מבוקרת של גני HOX בגידולים סרטניים. לדוגמה, נתיבי העברת האותות באמצעות תוצרי הגן Wnt ו-Hedgehog, שניהם גני HOX, מעורבים בהתפתחות סרטן המעי הגס וסוגי סרטן אחרים[12][13]. בנוסף נמצא שביטוי יתר של הגן Pax2 (אף הוא גן HOX) בגידולים סרטניים משפר את הישרדות תאי הגידול[7]. גני HOX נוספים שנמצאו שפעילותם דיכאה את ההתמיינות והגידול של תאים בגוף, כגון חלבון ה-HOX Msx1 המדכא את ההתמיינות של תאי אפיתל בעכבר[14].
גילוי גני ה-HOX
עריכהגני הוקס נקראים כך מכיוון שמוטציות בהם גורמות לטרנספורמציות הומיוטיות (אנ'), כלומר התפתחות אבולוציונות של איבר אחד לאיבר אחר. טרנספורמציות הומיוטיות זוהו ונחקרו לראשונה על ידי ויליאם בייטסון ב-1894, שטבע את המונח "הומאוזיס". לאחר הגילוי מחדש של העקרונות הגנטיים של מנדל, הבינו בייטסון ואחרים שניתן לייחס כמה דוגמאות של הומאוזיס באיברי פרחים ושלדי בעלי חיים לשונות בגנים.
גני HOX אופיינו לראשונה בתסיסנית המחקר כשנתגלה כי מוטציות בגנים ההומאוטיים הביאו לשינויים במיקום אברי הגוף[15]. לדוגמה, הגנים ההומאוטיים המהווים חלק ממכלול הגנים bithorax, אחראיים להתפתחות החלק האחורי בסגמנטי העובר של הזבוב[16][17][18]. גנים אלה נתגלו לאחר שבודדו מוטנטים של הדרוזופילה ולהם שני סגמנטי חזה. מכלול נוסף של גני HOX, שאחראי על התפתחות הסגמנטים הקדמיים של הדרוזופילה, נתגלה בעקבות בידוד מוטנט בו תוארה החלפה בין מיקום המחוש לבין הרגל השנייה בדרוזופילה[19][20]. התברר כי מקור המוטנט בפגם בגן Antennapedia, וההחלפה במיקום המחוש הייתה כבר אצל זבובים הטרוזיגוטיים לתכונה (+Antp/Antp)[21][22]. אנליזת רצף של הגן Antp הייתה הראשונה שהוליכה לגילוי ה-homeobox domain - מונח שנטבע לראשונה על ידי ולטר גרינג (אנ'). לאחר הגילוי הראשוני של גני HOX בדרוזופילה נתגלה שגנים אלו נמצאים גם בצפרדע Xenopus laevis - החולייתן הראשון בו תוארו הגנים[23][24][25].
מבנה ה-homeodomain
עריכהאזור ה-homeodomain מאפשר לחלבון ה-HOX להיקשר כפקטור שעתוק לרצף ספציפי באתרי בקרה של גנים המעורבים בתהליך ההתפתחות בעובר[26]. פעילות הקשירה שלו ל-DNA תוארה לראשונה ב-1994[25]. ה-homeobox מקודד לאזור ה homeodomain בגנים אלו[3]. למשל, בגן AntP של זבוב הדרוזופילה, אזור ה- homeodomain מורכב מ-4 סלילים (helix): סליל 1 הכולל את שיירי החומצות האמינות 10–21, לולאה בין חומצות אמינו 22–27 המקשרת את סליל 1 לסליל 2 (חומצות אמינו 28–38), סליל 3 מחומצות אמינו 42–52 והמתקשר לסליל 4 (חומצות אמינו 53-59)[25]. סליל 2 וסליל 3 יוצרים יחד מוטיב שנקרא helix-tum-helix שאופיין בעבר בחלבוני בקרת ביטוי גנים בחיידקים [27]. הקצה האמיני של חלבון זה מורכב מ־6 שיירים, 1-6, והוא חשוב מאוד לתהליך קשירת ה-DNA על ידי חלבון זה [28]. מבנה homeodomain זה דומה מאוד בגני HOX ביונקים וגם בחרקים[25].
ארגון גני ה-HOX בגנום
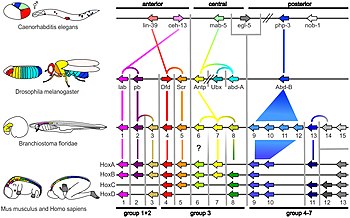
עריכהבאדם, חולדה, צפרדע ודג הזברה תוארו כ־200 גני HOX, בעוד שבחרקים, תולעים עגולות (נמטודות) ואזמלוניים יש כ-100 גנים[29][30].
מניתוחים של אילנות אבולוציוניים (פילוגנטיים) שנערכו עולה שמוצא גני ה-HOX בצמחים ובעלי חיים מורכבים הוא משני גנים קדומים[31]. בנוסף נמצא שגנים אלה מסודרים בגנום החולייתנים בצורה של אשכולות[32]. מבחינה אבולוציונית כל האשכולות של גנים אלה נוצרו מהכפלות של גנים מעטים ראשוניים[33].
הגנים ההומאוטיים בזבוב הדרוזופילה מסודרים בשני אשכולות - הראשון קומפלקס ה-Antennapedia וקומפלקס ה-Bithorax שהוא האחרון[34]. בחולייתנים יש 4 אשכולות HOX שבמקור נוצרו, כנראה, מהכפלה של אשכול אחד במהלך האבולוציה[35].
ניתן לחלק את גני ה-HOX למשפחות:
משפחת ה-Labial
עריכהמשפחת גנים אלו ממוקמת בקצה ה-'3 של אשכול HOX. גנים אלו בזבוב הדרוזופילה דומים ב-80% לגנים מקבוצה HOX1 בחולדה, ומראים 60% דמיון לשאר גני האשכול[36].
משפחת ה-Proboscipedia
עריכהמשפחה זו נמצאת בחולייתנים, ומכונה HOX2. קבוצת גנים זו דומה למשפחת הגנים בדרוזופילה, אך נעדרת בנמטודה – תולעים עגולות[37].
משפחת ה-Zerknüllt
עריכהגנים אלו בדרוזופילה שונים מגני HOX3 בחולייתנים, אך ניתוחים גנטיים בפרוקי-רגליים מציעים שגנים אלו בדרוזופוליה הומולוגיים לגני האשכול HOX3[38]. גנים אלה רכשו תפקידים חדשים במהלך האבולוציה, כמו קביעת דפוס של הציר גבי-גחוני במהלך ההתפתחות העוברית[39]. אחד מגנים אלו הוא ה-bicoid בדרוזופילה; נראה שגן זה הופיע כתוצאה מהכפלה של גן HOX3 במהלך האבולוציה[40].
משפחת ה-Deformed
עריכהמשפחה זו כוללת בתוכה את גן ה-Deformed בדרוזופילה וגן HOX4 של החולייתנים. משפחה זו אחראית להיווצרות הסגמנטים של הלסתות בראש של רימות זבוב הדרוזופילה[41].
משפחת ה-Sex-Combs-Reduced
עריכהמשפחה המכילה בתוכה הגן HOX5 והגן Scr בדרוזופילה. משפחה זו אחראית להתפתחות בית החזה והראש ברימות זבוב הדרוזופילה[42].
משפחת ה-Antennapedia
עריכהמשפחה המכילה בתוכה את הגנים הנחקרים ביותר באשכול: Antennapedia, Ultrabithorax, abdominal-A, fushi tarazu והגנים המקבילים (אורטולוגיים) מחולייתנים HOX6,HOX7 ו-HOX8. משפחה זו אחראית להתפתחות הכנפיים והרגליים בזבוב הדרוזופילה[43].
אשכול ה-Para-HOX
עריכהאשכול זה מורכב משלוש משפחות גנים: Gsx, Xlox ו-caudal האחרון[44]. הרצף של אשכול זה באזמלון דומה לרצף באשכול HOX, אך הסדר של הגנים שונה[45]. אשכול זה כנראה הופיע כתוצאה מהכפלה של HOX הקדמון. גנים אלו מקודדים לחלבונים בעלי תפקיד בקביעת הציר הקדמי-אחורי במהלך ההתפתחות העוברית[46].
אשכול ה-POU
עריכהקטגוריה זו מכילה 4 גנים: Pit-1, Oct-1, Oct-2 ו-Unc-86[47]. לגנים אלה יש תפקיד חשוב בהתפתחות מערכת העצבים המרכזית[48].
גני HOX באדם
עריכהגני HOX באדם מסודרים ב-4 אשכולות. עד עתה אופיינו כ-300 לוקוסים של גני HOX באדם, מתוכם 235 גנים פעילים ו-65 פסאודו-גנים[49].
סוגים נוספים של גני HOX
עריכהבנוסף לגני HOX שהוזכרו לעיל, יש עוד גני HOX שקשה לסווג אותם. לדוגמה, הגנים Hdx, Nobox ו-HopX שתוארו בחולייתנים - גנים אלה משחקים בין היתר תפקיד בהתפתחות הזקיקים בשחלה והתפתחות המערכת הלבבית באדם[50].
גני HOX בצמחים
עריכהבצמחים נמצאו 14 קטגוריות של גנים אלו, לצמח הארבידופסיס יש כ-100 גני HOX, ואנליזות מולקולריות הראו שרוב הגנים האלו גם מופיעים גם בבעלי חיים[51].
ניתוחים מולקולריים הראו שגנים אלה משחקים תפקיד חיוני בקביעת צורה וכיווניות של המרסטימה הקודקודית והפרחים[52].
הערות שוליים
עריכה- ^ Homeboxes, National Library of Medicine, 3 ביולי 2020
- ^ Peter W. H. Holland., Evolution of homeobox genes, Wiley Interdisciplinary Reviews-Developmental Biology, 2012, עמ' 31-45
- ^ 1 2 Bürglin TR., Affolter M., Homeodomain proteins: an update, Chromosoma 125, 2016, עמ' 497–521
- ^ המושג Homeo מקורו מהמילה היוונית hómoios - ומשמעותו, במסגרת מחקר הבסיס המולקולרי של ההתפתחות העוברית, יצירת מבנה מסוים במקום מסוים (סגמנט) בגוף העובר.
- ^ Bürglin TR., Homeodomain Subtypes and Functional Diversity, Sub-cellular biochemistry 52, 2011, עמ' 95-122
- ^ Young RA, Control of the embryonic stem cell state, Cell 144, 2011, עמ' 940- 954
- ^ 1 2 S. Samuel, H. Naora, Homeobox gene expression in cancer: Insights fromdevelopmental regulation and deregulation, European Journal of Cancer 41, 2005, עמ' 2428–2437
- ^ Driever W, Nüsslein-Volhard C, The bicoid protein determines position in the Drosophila embryo in a concentration-dependent manner, Cell 54, 1988, עמ' 95-104
- ^ Brandt R, Cabedo M, Xie Y, Wenkel S, Homeodomain leucine-zipper proteins and their role in synchronizing growth and development with the environment, J Integr Plant Biol 56, 2014, עמ' 518-526
- ^ Costanzo E, Trehin C, Vandenbussche M, The role of WOX genes in flower development, Ann Bot 114, 2014, עמ' 1545-1553
- ^ Tsuda K, Hake S, Diverse functions of KNOX transcription factors in the diploid body plan of plants, Curr Opin Plant Biol 27, 2015, עמ' 91-96
- ^ J. Taipale and P.A. Beachy, The Hedgehog and Wnt signalling pathways in cancer, Nature 411, 2001, עמ' 349-354
- ^ T. Reya and H. Clevers, Wnt signalling in stem cells and cancer, Nature 434, 2005, עמ' 843-850
- ^ G. Hu, H. Lee, S.M. Price, Msx homeobox genes inhibit differentiation through upregulation of cyclin D1, Development 128, 2001, עמ' 2373-2384
- ^ Bateson, W, Materrals for the Study of Variation, London: MacMillan and Co, 1894
- ^ Lewis,E.B, Genes and developmental pathways, Am. Zool. 3, 1963, עמ' 33-56
- ^ Lewis,E.B, A gene complex controlling segmentation in Drosophila, Nature 276, 1978, עמ' 565-570
- ^ Duncan,I. and Lewis,E.B, Developmental Order: Its Origin and Regulation, NY: A.R.Liss, 1982, עמ' 533-554
- ^ Lewis,R.A., Kaufman,T.C., Denell,R.E. and Tallerico,P, Lewis,R.A., Kaufman,T.C., Denell,R.E. and Tallerico,P, , Genetics 95, 1980, Genetics 95, 1980, עמ' 367-381
- ^ Kaufman,T.C., Lewis,R. and Wakimoto,B., Cytogenetic Analysis of Chromosome 3 in DROSOPHILA MELANOGASTER: The Homoeotic Gene Complex in Polytene Chromosome Interval 84a-B, Genetics 94, 1980, עמ' 115-133
- ^ Lewis,E.B, Drosophila Inf, Inf. Serv, 1956, עמ' 76
- ^ Falk,R, Drosophila Inf, Serv, 1964, עמ' 60
- ^ McGinnis W, Levine MS, Hafen E, Kuroiwa A, Gehring WJ, A conserved DNA sequence in homoeotic genes of the Drosophila Antennapedia and bithorax complexes, Nature 308, 1984b, עמ' 428-433
- ^ Carrasco AE, McGinnis W, Gehring WJ, De Robertis EM, Cloning of an X. laevis gene expressed during early embryogenesis coding for a peptide region homologous to Drosophila homeotic genes, Cell 37, 1984, עמ' 409-414
- ^ 1 2 3 4 W. J. Gehring, M. Affolter, T. Bürglin, Homeodomain proteins, Annual Review of Biochemistry 63, 1994, עמ' 487-526
- ^ H. Mihara, E.T. Kaiser, A chemically synthesized Antennapedia homeo domain binds to a specific DNA sequence, Science 242, 1988, עמ' 925-927
- ^ C. O. Pabo, R. T. Sauer, Transcription Factors: Structural Families and Principles of DNA Recognition, Annual Review of Biochemistry 61, 1992, עמ' 1053-1095
- ^ W. J. Gehring, Y. Q. Qian, M. Billeter, K. Furukubo-Tokunaga, A. F.Schier, D. Resendez-Perez, M. Affolter, G. Otting, K. Wüthrich, Homeodomain-DNA recognition, Cell 78, 1994, עמ' 211-223
- ^ Zhong Y-F, Butts T, Holland PWH, HomeoDB: adatabase of homeobox gene diversity, Evol Dev 10, 2008, עמ' 516–518
- ^ Zhong Y-F, Holland PWH, HomeoDB2: functional expansion of a comparative homeobox gene databasefor evolutionary developmental biology, Evol Dev 13, 2011, עמ' 567–568
- ^ Bharathan G, Janssen BJ, Kellogg EA, Sinha N, Did homeodomain proteins duplicate before the origin of angiosperms, fungi, and metazoa?, Proceedings of the National Academy of Sciences of the United States of America 94, 1997, עמ' 49-53
- ^ Frank H. Ruddle, Janet L. Bartels, Kevin L. Bentley, Claudia Kappen1, Michael T. Murtha , and John W. Pendleton, Evolution of HOX genes, Annu. Rev. Genet 28, 1994, עמ' 23-42
- ^ Garcia-Fernàndez J, The genesis and evolution of homeobox gene clusters, Nature Reviews Genetics 6, 2005, עמ' 81-92
- ^ Duboule D, Morata G, Colinearity and functional hierarchy among genes of the homeotic complexes, Trends Genet 10, 1994, עמ' 358–364
- ^ Crow KD, Stadler PF, Lynch VJ, Amemiya C, Wagner GP, The “fish-specific” Hox cluster duplication is coincident with the origin of teleosts, Mol Biol Evol 23, 2006, עמ' 121-136
- ^ Kaufman, T. C., Seeger, M. A. & Olsen, G, Molecular and genetic organization of the antennapedia gene complex of Drosophila melanogaster, Adv. Genet 27, 1990, עמ' 309-362
- ^ Hunter CP, Kenyon C, Specification of anteroposterior cell fates in Caenorhabditis elegans by Drosophila Hox proteins, Nature 377, 1995, עמ' 229-32
- ^ Damen WGM, Tautz D, A Hox class 3 orthologue from the spider Cupiennius salei is expressed in a Hox-gene-like fashion, Dev Genes 208, 1998, עמ' 586-590
- ^ Stauber M, Jäckle H, Schmidt-Ott U, The anterior determinant bicoid of Drosophila is a derived Hox class 3 gene, Proc Natl Acad Sci USA 96, 1999, עמ' 3786–3789
- ^ Stauber M, Prell A, Schmidt-Ott U, A single Hox3 gene with composite bicoid and zerknüllt expression characteristics in non-Cyclorrhaphan flies, . Proc Natl Acad Sci USA 99, 2002, עמ' 274-279
- ^ Krumlauf, R, Hox genes in vertebrate development, Cell 78, 1994, עמ' 191-201
- ^ Brooke, N.M., Garcia-Fernandez, J., Holland, P.W.H., The ParaHox gene cluster is an evolutionary sister of the Hox gene cluster, Nature 392, 1998, עמ' 920-922
- ^ Dawes R, Dawson I, Falciani F, Tear G, Akam M, Dax, a locust Hox gene related to fushi-tarazu but showing no pair-rule expression, Development 120, 1994, עמ' 1561-1572
- ^ Ferrier DE, Minguillon C, Evolution of the Hox/ParaHox gene clusters, Int J Dev Biol 47, 2003, עמ' 605-611
- ^ Brooke NM, Garcia-Fernàndez J, Holland PWH, The ParaHox gene cluster is an evolutionary sister of the Hox gene cluster, Nature 392, 1998, עמ' 920-922
- ^ Garcia-Fernàndez J, The genesis and evolution of homeobox gene clusters, Nat Rev Genet 6, 2005, עמ' 811-892
- ^ Herr W, Sturm RA, Clerc RG, Corcoran LM, Baltimore D, Sharp PA, Ingraham HA, Rosenfeld MG, Finney M, Ruvkun G, et al, The POU domain: a large conserved region in the mammalian pit-1, oct-1, oct-2, and Caenorhabditis elegans unc-86 gene products, Genes Dev 2, 1988, עמ' 1513-1516
- ^ R. Josephson, T. Müller, J. Pickel, S. Okabe, K. Reynolds, P. A. Turner, A. Zimmer and R. D. G. McKay, POU transcription factors control expression of CNS stem cell-specific genes, Development 125, 1998, עמ' 3087-3100
- ^ P. WH Holland, H. Anne F Booth, E. A. Bruford, Classification and nomenclature of all human homeobox genes, BMC Biol. 5, 2007, עמ' 47
- ^ Kagoshima H, Cassata G, Bürglin TR, A Caenorhabditis elegans homeobox gene expressed in the male tail, a link between pattern formation and sexual dimophism?, Dev Genes Evol 209, 1999, עמ' 59-62
- ^ Mukherjee K, Brocchieri L, Bürglin TR, A comprehensive classification and evolutionary analysis of plant homeobox genes, Mol Biol Evol 26, 2009, עמ' 2775-2794
- ^ R. W. Williams, Plant homeobox genes: many functions stem from a common motif, Bioessays, 1998, עמ' 280-2