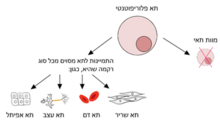
ייעוד התא
המושג "ייעוד התא" מתייחס לזהות העתידית או לתוצאה ההתפתחותית של תא בתוך אורגניזם. הוא מתאר סוג התאים הספציפי שתא מסוים ישתייך אליו לאחר שיעבור תהליכי התמיינות. קביעת ייעוד התא הינו תחום בסיסי בביולוגיה התפתחותית והוא קובע, כי במהלך ההתפתחות העוברית, תאים מסוימים יתמיינו לסוגי תאים מסוימים, ומאוחר יותר לאיברים מסוימים בגוף העובר המתפתח (ראו איור 1).[1]
בתוך עובר מתרחשים מספר תהליכים אשר קובעים את תבנית ההתפתחות של האורגניזם כולו. תהליכים אלה כוללים גדילה והתרבות של תאים, תנועת תאים, התמיינות תאים ועוד[1]. כל תא בעובר המתפתח מקבל אותות מתאים שכנים, הכוללים בין היתר חלבונים, ומתנאי הסביבה (חומציות למשל). כמעט כל בעלי החיים עוברים רצף אירועים דומה בתחילת התהליך של התפתחות העובר (embryogenesis). רצף אירועים זה שמור מבחינה אבולוציונית[2][3]. חוקרים מצאו שישנם חלבונים ומולקולות mRNA מסוימים וקבועים, המעורבים בתהליך ההתפתחות[4]. העובדה שתהליך זה שמור מבחינה אבולוציונית מאפשרת שימוש בחיות מודל שונות (כמו למשל הזבוב תסיסנית המחקר) לצורך הסקת מסקנות ותובנות הרלוונטיות לבעלי חיים אחרים, לרבות בני אדם.
נמצא שייעוד התא נקבע במספר דרכים, ובהן שילוב בין גורמי תעתוק הקיימים בתא עצמו לבין חלבונים המעורבים באינטראקציות בין תאיות[5]. את קביעת ייעוד התא ניתן לחלק לשני שלבים:
- specification - שלב זה חל מוקדם יותר, ובו התא מצוי בתהליכי התחייבות לקראת הפיכתו לתא מסוג מסוים, על אף שלא נצפו בו עדיין סממנים חיצוניים לכך. שלב זה הפיך, ומושפע לרוב מגורמים גנטיים ומאותות שהתא מקבל מסביבתו.
- determination - שלב זה חל מאוחר יותר, וכולל בתוכו את התפתחות המאפיינים והפונקציות הספציפיים לאותו סוג תא. הוא אינו הפיך, ובמהלכו התא עובר שינויים מורפולוגיים ותפקודיים אשר יובילו בסופו של דבר להיווצרות רקמות ואיברים שונים[6].
המנגנון העומד בבסיס קביעת ייעוד התאים
עריכהבביולוגיה התפתחותית, התמיינות התאים נשלטת על ידי רשת בקרת תאים (gene regulatory network – GRN)[7], וכוללת גורמי תיעתוק המבקרים את תהליך ההתמיינות. חלוקת תאי העובר ותהליך ההתמיינות שונה בין בעלי החיים השונים, אך עם זאת, ניתן למצוא עקרונות GRN דומים בין בעלי חיים שונים; אלה מצדיקים את השימוש בחיות מודל כדי להעריך את הכלליות של התופעה. מעקב אחרי תהליך ההתפתחות בחיות מודל מאפשר מיפוי גורל (אנ'), תוך צביעה של תאי אם (למשל בפלואורסצנציה) ומעקב אחר תהליכי ההתפתחות העוברית וחלוקת התאים עד לקבלת תא ממוין, או מאוחר יותר - איבר[8][9]. גישה זו יעילה למעקב אחר התמיינות תאים, אך היא תיאורית בלבד (descriptive) ואינה מספקת הסבר למנגנונים המעורבים בקביעת ייעוד התא. על מנת להבין מנגנונים אלה, יש צורך בשילוב בין מיפוי ייעוד התאים לבין שיטות מולקולריות נוספות, בהן מתערבים בביטוי של גנים המעורבים בקביעת ייעוד התאים - שיטות כגון השתקת גנים (knock-down) או ביטוי יתר של גנים (over-expression) בעובר.
בתחילת תהליך התפתחות העובר, למשך מספר חלוקות (המספר משתנה בין אורגניזמים שונים), התאים הם פלוריפוטנטיים (אנ') - כלומר, מסוגלים להתמיין לכל סוג תא.במילים אחרות, לתאים שונים יש אותו פוטנציאל התמיינותי[10]. התקדמות תהליך ההתפתחות העוברי מלווה במספר גדל והולך של גורמים המשפיעים על התמיינות התא: גורמים המעורבים באינדוקציה חוץ תאית ובאינטראקציה בין תאית; נוכחות גורמי תעתוק מסוימים ו-mRNAs וכדומה. לדוגמה, אינדוקציה חוץ תאית – נבדקה במערכת "P-O" (קבוצת תאים בעלי פוטנציאל התמיינות זהה) בעלוקה Helobdella (אנ'), ונמצא שאותות מתאים שכנים משפיעים על ייעוד תאי O/P3. במערכת ה-"P-O" נבחנה גם אינטראקציה בין תאית, כלומר אינטראקציה המתקיימת בתוך אותה מערכת, בין התאים בעלי פוטנציאל ההתמיינות הזהה לבין עצמם. חשיבות גורמי התעתוק נבדקה בין היתר במין תסיסנית המחקר, המשמש לעיתים קרובות כחיית מודל. מצאו למשל שגורם תעתוק בשם "Dorsal" המייצר גרדיאנט ביטוי של גורמי תיעתוק לאורך הציר הבטני-גבי (דורסלי-ונטרלי) וכך למעשה משפיע על ייעוד התא לאורך ציר זה[11].
בעבר לא ניתן היה להחזיר את מצב התאים ולהופכם לבלתי ממוינים. מחקר שנערך בשנים האחרונות גילה שקיימת אפשרות לגרום ל"איבוד" ההתמיינות, כלומר להחזיר תאים לנקודת ההתחלה, במצבם הבלתי ממוין, גם לאחר שכבר התמיינו[12][13][14]. במחקר זה נתגלה כי די בביטוי ארבעה גורמי תעתוק (Oct3/4, Sox2, Klf4, and c-Myc) על מנת להפוך תאים סומטיים ממוינים בתרבית לתאי גזע בעלי יכולת התמיינות לכל סוג תא – induced pluripotent stem cells (iPSCs). עוד נתגלה במחקר זה, שתאים ממוינים אשר הושרה בהם ביטוי יתר של ארבעת גורמי התעתוק המוזכרים לעיל, הציגו מורפולוגיה ותכונות המאפיינות תאי עובר, כולל ביטוי של סמנים (גנים) של תאים פלוריפוטנטיים. בנוסף, השתלה תת-עורית של תאי iPS לעכברים הביאה ליצירת גידולים המכילים תאים המאפיינים מגוון רקמות; כך, נוצר גידול המכיל רקמת שריר, נוירונים ואפיתל[14]. ממצא זה מדגים גם את פוטנציאל ההתמיינות של תאי iPS וגם מדגים את הקשר בין התמיינות לבין התהליך הסרטני.
תהליך התחייבות התא לייעוד מסוים עשויה לתקיים באמצעות שלושה מנגנונים שונים:
- סציפיקציה אוטונומית (Autonomous Specification) – במנגנון מסוג זה, אם נבודד בלסטומר[15] ונגדל אותו בנפרד, הוא ייצר את אותם התאים שהיה מייצר לו עדיין היה חלק מהעובר. כמו כן, בעובר ממנו נלקח הבלסטומר, יחסרו אותם התאים אשר היו אמורים להיות מיוצרים על ידי הבלסטומר.
- ספציפיקציה מותנית (Conditional specification) – במנגנון מסוג זה, ייעוד התא תלוי באינטראקציות שהוא יוצר עם הסביבה ובתנאים בהם הוא נמצא. אם נוציא בלסטומר במצב זה מעובר, התאים העובריים הנותרים יוכלו לשנות את ייעודם, ועל ידי כך למלא את החסר.
- ספציפיקציית סינקיטיון (Syncytial specification) – במצב זה, הגרעין מתחלק והציטופלזמה נותרת מוקפת בממברנת התא ללא חלוקה. תא כזה, שלו גרעינים מרובים, נקרא סינקיטיון. מצב זה מאפשר תהליכי התמיינות בתוך הסינקיטיון לקביעת הייעוד של גרעינים, כדי שיהוו מקור להתמיינות לסוגי תאים ואיברים. החלבונים הקובעים את ייעוד הגרעינים נקראים מורפוגניים. הם משפיעים על גרעיני התא לפי ריכוזם ברחבי הסינקיטיון. זהו המצב בראשית ההתפתחות העוברית בחרקים.
שינויים מורפולוגיים בתהליך קביעת ייעוד התא
עריכהבתהליך קביעת ייעוד התא מתרחשים שינויים מורפולוגיים הנקבעים בהתאם לסוג התא. לדוגמה, בשלב הגסטרולציה של העובר, תאי אפיתל עמודי (אנ') ישנו צורה לתאים מזנכימליים, שביחס לתאי האפיתל העמודי, הם ארוכים יותר ומאופיינים באחיזה חלשה יותר[16]. בנוסף, עשויים ללוות את התהליך שינויים בממברנת התא (ובפרט בדינמיות שלה), ובכלל זאת היווצרות של אזורים בממברנה היוצרים "מתחם", או שינויים ביכולת ההיצמדות לתא אחר[17].
מעורבות אפיגנטיקה בקביעת ייעוד התאים
עריכההאפיגנטיקה משחקת גם היא תפקיד חיוני בבקרת תהליך קביעת ייעוד התא בהתפתחות העובר. שינויים אפיגנטיים בגנום מווסתים ביטוי גנים מבלי לשנות את רצף ה-DNA ומאפשרים שליטה דינמית על ביטוי אותם הגנים. כאמור, תהליך קביעת ייעוד התא מושפע בין היתר מביטוי גנים ספציפיים, בהתאם לסוג התא. בתהליך קביעת ייעוד התא מעורבים מספר שינויים אפיגנטיים, ביניהם –
- מתילציה של DNA – מתילציית DNA, במיוחד באתרי CpG, היא סימן אפיגנטי לדיכוי ביטוי גנים. בשלבים המוקדמים של התפתחות העובר מתרחשת דה-מתילציה לאורך כל הגנום ולאחריה ביסוס דפוסי מתילציה ספציפיים[18][19].
- שינויים בהיסטונים – מתילאציה ואצטילאציה - שינויים המתרחשים בהיסטונים לאחר תרגום עלולים להשפיע על מבנה הכרומטין אותו הם קשרו. כתוצאה מכך, נגישות גנים בגנום משתנה וכך גם תשתנה תבנית הביטוי שלהם[20]. מתילציה – תהליך בו מתרחשת הוספת קבוצת מתיל (3CH) למולקולת סובסטרט, או החלפת אטום/מולקולה על ידי קבוצת מתיל. אצטילציה – תהליך בו מתרחשת הוספת קבוצת אצטיל, בעיקר על השייר האמיני (3NH+) של חומצת האמינו ליזין בחלבונים.
גורמים נוספים המשפיעים על קביעת ייעוד התא
עריכהמחקרים מצאו כי בנוסף לתהליכים המולקולריים המוזכרים לעיל ישנם גורמים נוספים המשפיעים על תהליך ההתמיינות. בין גורמים אלו נמנים שינויים מטבוליים, שינוי בתבנית העברת האותות בתא דרך מטבוליטים ורדיקלים חופשיים של חמצן - מיני חמצן ראקטיביים (אנ') (ROS), pH תוך תאי, מורפולוגיה של התא ואינטראקציה עם התווך החוץ תאי[21].
שיטות מחקר
עריכההשימוש בנמטודה Caenorhabditis elegans בחקר ייעוד התא נפוץ מאוד. הסיבה לכך, היא, שלמרות שמדובר באורגניזם רב תאי, נמטודה זו פשוטה יחסית לצורכי מחקר. היא בעלת מספר קבוע של תאים (959 בתולעת הרמפרודיטית – בעלת אברי מין זכריים ונקביים גם יחד)[22], הגנום שלה רוצף בשלמותו, מבנה הגוף שלה סימטרי, ותהליך התפתחותה נמשך כשלושה ימים בסך הכול. דפוס חלוקת האתים והתמיינותם בנמטודה זו קבוע מפרט לפרט, ועל כן ניתן לחזות את ייעודו של כל תא לפי מיקומו ב"אילן" התמיינות התאים[22]. השימוש בחלבון פלואורסצנטי ירוק (GFP), שמקורו במדוזה Aequorea victoria (אנ'), אשר בעת הקרנתו תחת אור כחול או אולטרה סגול מגיב בפלואורוצנציה, מאפשר תיוג של חלבונים ותאים על ידי החלבון הפלואורסצנטי הירוק ומעקב אחר ביטויים ומיקומם של חלבונים ותאים אלה לאורך תהליך ההתמיינות.
התקדמות בשיטות מעקב ומניפולציה על גנום של חיות מודל מוסיף עומק לאפשרויות המחקר. לדוגמה, השימוש ב-Single-cell RNA Sequencing (scRNA-seq) מספק פרופיל ביטוי גנים של תאים בודדים. בזכות כך, הוא מאפשר זיהוי של סוגים ומצבים שונים של תאים על פי דגם ביטוי גנים בכל הגנום, וכן שחזור של מסלול התפתחות התא, והבחנה בין אוכלוסיות תאים שונות[23]. כמו כן, מניפולציות גנטיות הנערכות באמצעות מערכת קריספר מאפשרות מחקר אודות תפקיד של גנים. בנוסף, ניתן להחדיר גן דווח לתוך לוקוסים מסוימים בגנום, ועל ידי כך לעקוב אחר שושלת תאים תוך כדי התפתחות העובר, ולהגיע לתובנות אודות ייעוד התא[24].
לקריאה נוספת
עריכה- Casey, M. J., Stumpf, P. S., & MacArthur, B. D. (2020). Theory of cell fate. Wiley Interdisciplinary Reviews: Systems Biology and Medicine, 12(2), e1471.
- Wagers, A. J., Christensen, J. L., & Weissman, I. L. (2002). Cell fate determination from stem cells. Gene therapy, 9(10), 606-612.
- Iwafuchi-Doi, M., & Zaret, K. S. (2016). Cell fate control by pioneer transcription factors. Development, 143(11), 1833-1837.
הערות שוליים
עריכה- ^ 1 2 John B Wallingford, Scott E Fraser, Richard M Harland, Convergent Extension, Developmental Cell 2, 2002-06, עמ' 695–706 doi: 10.1016/s1534-5807(02)00197-1
- ^ Suzanne V Saenko, Vernon French, Paul M Brakefield, Patrícia Beldade, Conserved developmental processes and the formation of evolutionary novelties: examples from butterfly wings, Philosophical Transactions of the Royal Society B: Biological Sciences 363, 2008-01-11, עמ' 1549–1556 doi: 10.1098/rstb.2007.2245
- ^ Alex T. Kalinka, Pavel Tomancak, The evolution of early animal embryos: conservation or divergence?, Trends in Ecology & Evolution 27, 2012-07, עמ' 385–393 doi: 10.1016/j.tree.2012.03.007
- ^ Michael Levine, Eric H. Davidson, Gene regulatory networks for development, Proceedings of the National Academy of Sciences 102, 2005-03-23, עמ' 4936–4942 doi: 10.1073/pnas.0408031102
- ^ Charles H. Streuli, Integrins and cell-fate determination, Journal of Cell Science 122, 2009-01-15, עמ' 171–177 doi: 10.1242/jcs.018945
- ^ 1 2 Daniela Palacios, Pier Lorenzo Puri, Switch NFix Developmental Myogenesis, Developmental Cell 18, 2010-03, עמ' 340–341 doi: 10.1016/j.devcel.2010.03.005
- ^ Morris F. Maduro, Cell fate specification in the C. elegans embryo, Developmental Dynamics 239, 2010-01-27, עמ' 1315–1329 doi: 10.1002/dvdy.22233
- ^ Steffen Wolf, Yinan Wan, Katie McDole, Current approaches to fate mapping and lineage tracing using image data, Development 148, 2021-09-09 doi: 10.1242/dev.198994
- ^ Emilie Legué, Alexandra L. Joyner, Genetic Fate Mapping Using Site-Specific Recombinases, Elsevier, 2010, עמ' 153–181, ISBN 978-0-12-384880-2
- ^ Françoise Z. Huang, David A. Weisblat, Cell fate determination in an annelid equivalence group, Development 122, 1996-06-01, עמ' 1839–1847 doi: 10.1242/dev.122.6.1839
- ^ N. Perrimon, C. Pitsouli, B.-Z. Shilo, Signaling Mechanisms Controlling Cell Fate and Embryonic Patterning, Cold Spring Harbor Perspectives in Biology 4, 2012-08-01, עמ' a005975–a005975 doi: 10.1101/cshperspect.a005975
- ^ Kazutoshi Takahashi, Koji Tanabe, Mari Ohnuki, Megumi Narita, Tomoko Ichisaka, Kiichiro Tomoda, Shinya Yamanaka, Induction of Pluripotent Stem Cells from Adult Human Fibroblasts by Defined Factors, Cell 131, 2007-11, עמ' 861–872 doi: 10.1016/j.cell.2007.11.019
- ^ Junying Yu, Maxim A. Vodyanik, Kim Smuga-Otto, Jessica Antosiewicz-Bourget, Jennifer L. Frane, Shulan Tian, Jeff Nie, Gudrun A. Jonsdottir, Victor Ruotti, Ron Stewart, Igor I. Slukvin, James A. Thomson, Induced Pluripotent Stem Cell Lines Derived from Human Somatic Cells, Science 318, 2007-12-21, עמ' 1917–1920 doi: 10.1126/science.1151526
- ^ 1 2 Kazutoshi Takahashi Shinya Yamanaka, Induction of Pluripotent Stem Cells from Mouse Embryonic and Adult Fibroblast Cultures by Defined Factors, Cell 4, עמ' 663-676
- ^ גופיף עוברי המורכב מתאים המסוגלים להתמיין אך ורק לתאים מסוימים.
- ^ Marta N. Shahbazi, Mechanisms of human embryo development: from cell fate to tissue shape and back, Development 147, 2020-07-15 doi: 10.1242/dev.190629
- ^ Chii J. Chan, Carl-Philipp Heisenberg, Takashi Hiiragi, Coordination of Morphogenesis and Cell-Fate Specification in Development, Current Biology 27, 2017-09, עמ' R1024–R1035 doi: 10.1016/j.cub.2017.07.010
- ^ Lionel A Sanz, Satya K Kota, Robert Feil, Genome-wide DNA demethylation in mammals, Genome Biology 11, 2010, עמ' 110 doi: 10.1186/gb-2010-11-3-110
- ^ T. Haaf, Methylation Dynamics in the Early Mammalian Embryo: Implications of Genome Reprogramming Defects for Development, Springer Berlin Heidelberg, עמ' 13–22, ISBN 978-3-540-31180-5
- ^ Chuan Chen, Yawei Gao, Wenqiang Liu, Shaorong Gao, Epigenetic regulation of cell fate transition: learning from early embryo development and somatic cell reprogramming, Biology of Reproduction 107, 2022-05-07, עמ' 183–195 doi: 10.1093/biolre/ioac087
- ^ Sumitra Tatapudy, Francesca Aloisio, Diane Barber, Todd Nystul, Cell fate decisions: emerging roles for metabolic signals and cell morphology, EMBO reports 18, 2017-11-20, עמ' 2105–2118 doi: 10.15252/embr.201744816
- ^ 1 2 Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, Peter Walter, Molecular Biology of the Cell, 2007-12-31 doi: 10.1201/9780203833445
- ^ Aleksandra A. Kolodziejczyk, Jong Kyoung Kim, Valentine Svensson, John C. Marioni, Sarah A. Teichmann, The Technology and Biology of Single-Cell RNA Sequencing, Molecular Cell 58, 2015-05, עמ' 610–620 doi: 10.1016/j.molcel.2015.04.005
- ^ Mingze Yao, Tinglin Ren, Yuanqing Pan, Xiaoqing Xue, Rong Li, Lei Zhang, Yuhang Li, Ke Huang, A New Generation of Lineage Tracing Dynamically Records Cell Fate Choices, International Journal of Molecular Sciences 23, 2022-04-30, עמ' 5021 doi: 10.3390/ijms23095021