מוטגנזה מכוונת למיקום מדויק
מוטגנזה מכוונת למיקום מדויק (Site directed mutagenesis) היא שיטה בביולוגיה המולקולרית שמשמשת ליצור שינויים (מוטציות) מכוונים ברצף ה-DNA. הטכניקה הזו מאפשרת לחקור את המבנה והפונקציה של גנים וחלבונים באמצעות שינוי מדויק וספציפי של הקוד הגנטי שלהם. כך ניתן לחקור את ההשפעה של שינוי חומצות אמינו ספציפיות על הפעילות של חלבונים. שינוי מכוון ביותר מגן אחד יכול לשמש להנדסה גנטית של תאים בתרבית, ובכך לאפשר מחקרים יישומיים וביוטכנולוגים.
תהליך המוטגנזה המכוונת מתחיל בעיצוב מקטע DNA קצר הנקרא אוליגונוקלאוטיד (אנ'), אשר מתאים לרצף המטרה אבל מכיל בתוכו את המוטציה הרצויה[1][2]. אוליגונוקלאוטיד זה מתחבר ל-DNA חד-גדילי של הגן המיועד על פי עיקרון התאמת הבסיסים (A ל-T, G ל-C), ונוצרת מולקולה דו-גדילית, שאחד מגדיליה מכיל את הרצף המקורי, ואילו הגדיל השני שלה מכיל את האוליגונוקלאוטיד עם המוטציה. לאחר שהאוליגונוקלאוטיד מתחבר ל-DNA פולימראז שמאריך אותו, מסונתז גדיל DNA חדש המכיל את המוטציה. זה מוליך ליצירת מולקולת DNA דו-גדילית שבה גדיל אחד הוא המקורי והגדיל השני הוא המוטנטי. לדוגמה, אם המוטציה נעשתה באופן מכוון ברצף חיידקי, ה-DNA המוטנטי מוחדר לתוך תאי חיידקים שמתקיימים בתוכו תהליכי שכפול DNA. במהלך השכפול, אם המוטציה אינה מתוקנת, היא תשמש כתבנית, ותשתכפל שוב ושוב עד למצב בו שני גדילים מכילים את המוטציה הרצויה. כך ניתן לבודד מושבות חיידקים עם DNA מוטנטי בלבד (שיבוט) ולרצף את ה-DNA כדי לוודא את קיום המוטציה (איור 1). אם המוטציה המתוכננת אמורה לגרום לשינוי פנוטיפי בחיידק – ניתן יהיה לבדוק אותו בניסוי.
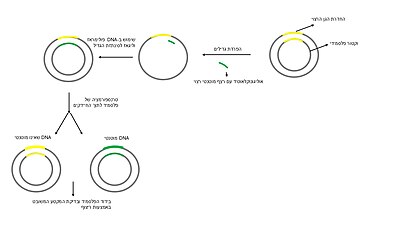
באיאוקריוטיים, התהליך של יצירת המוטציות מתקיים במהלך חלוקה תאית. אחרי הכנסת ה-DNA המוטנטי לתא, התא יכול לעבור תהליכי תיקון DNA ולאחר מכן רקומבינציה הומולוגית, שבמהלכה הגדיל המקורי מוחלף בגדיל המכיל את המוטציה. כתוצאה מרקומבינציה הומולוגית, נוצרות כרומטידות אחיות, שכל אחת מהן מכילה את המוטציה. במהלך שלב המיטוזה, הכרומטידות האחיות נפרדות, וכל אחת מהן עוברת לתא-בת. אם שני תאי-הבת מקבלים את הכרומטידות המוטנטיות, שניהם יכילו רצף מוטנטי. כדי להגיע למצב הומוזיגוטי צריך לקרות תהליך של המרת גן (אנ') (Gene conversion). בתהליך זה, מקטע DNA מכרומטידה של אחד מהכרומוזומים ההומולוגיים "פולש" אל הסליל הכפול של הכרומוזום ההומולוגי, והרצף של הכרומוזום ההומולוגי מתוקן על פי הרצף ה"פולש". תהליך זה גורם לרצף השני להכיל את המוטציה באופן זהה לרצף שפלש. כתוצאה מכך, אם התאים שבהם התרחש תהליך המרת הגן (Gene conversion) ימשיכו להתחלק, תיווצר אוכלוסייה של תאים הומוזיגוטים למוטציה[3][4][2].
ההיסטוריה של מוטגנזה מכוונת למיקום מדויק
עריכהמוטגנזה מכוונת למיקום מדויק תוארה לראשונה על ידי מייקל סמית' בשיתוף פעולה עם קלייד האצ'יסון בסוף שנות ה-70 של המאה ה-20, מתוך כוונה ליצור מוטציות ספציפיות ברצף ה-DNA[4]. בשנת 1978, הם פרסמו מאמר שמתאר את השיטה שלהם על הכנסת מוטציות נקודתיות ל-DNA פלסמידי באמצעות אוליגונוקלאוטיד (oligonucleotide): החוקרים ביצעו מוטציה בגן E בפאג' -φX174. החוקרים החליפו את הנוקלאוטיד גואנין (G) בעמדה 587 לאדנין (A) כדי ליצור את המוטנט am3 בגן E. אחת מהבעיות העיקריות של הטכניקה הייתה היעילות הנמוכה של הכנסת המוטציות הרצויות. בנוסף, היה קושי בזיהוי של גדילים מוטנטיים אחרי תהליך השכפול[4].
בשנת 1985, תומאס א. קונקל הציג טכניקה חדשה למוטגנזה מכוונת למיקום מדויק ששיפרה את יעילות הכנסת המוטציות. שיטה זו כוללת הכנסת מקטע DNA מוטנטי, ואחר כך העברתו לזן של חיידק E. coli, שחסרים לו שני אנזימים: dUTPase ו-uracil deglycosidase, שהם חלק במסלול תיקון ובקרה של ה-DNA, שתפקידו הגנה על הכרומוזום הבקטריאלי מפני מוטציות. חוסר באנזימים הללו גרם לייצור רמות גבוהות של dUTP ב־E. coli, וזה הוביל, בתורו, לשילוב האורציל ב־DNA חד גדילי. מקטע זה שימש כתבנית למוטגנזה בעזרת אוליגונוקלאוטיד-oligonucleotide שמכיל את המוטציה הרצויה. לאחר מכן נוצר גדיל אחד ללא מוטציה, שהכיל dUTP, והגדיל השני התקיים עם מוטציה והכיל dTTP. שיפורים אלה הפכו את המוטאגנזה המכוונת לשיטה מדויקת ויעילה ביותר ליצירת מוטציות ספציפיות ברצף DNA לפי בחירה[5][6].
מאז פיתוח המוטגנזה המכוונת בשנות ה-70 וה-80 של המאה ה-20, פותחו ושוכללו שיטות רבות בתחום זה. הטכניקות המודרניות מתבססות על עקרונות הניסויים הראשונים, אך שופרו והותאמו לצרכים השונים ולשיפור היעילות והדיוק. בשנת 2014, פותחה גם השיטה CRISPR/Cas9 להחדרת מוטציות מדויקות, ואפילו שיטה ל-Knock out של גנים[7][8].
שיטות מבוססות על מוטגנזה מכוונת למיקום מדויק
עריכהבשיטה הקלאסית, משתמשים באוליגונוקלאוטיד (פריימר) מוטנטי ובשני פריימרים אוניברסליים. בשלב ראשון מתבצע PCR תוך שימוש בפריימר המוטנטי ובאחד מהפריימרים האוניברסליים. ביחד הם יוצרים מגה-פריימר (megaprimer) שבו משתמשים לראקציית PCR שנייה יחד עם פריימר אוניברסלי נוסף. ניתן להשתמש בתוצר שמכיל את המוטציה כדי להכניס אותה באמצעות שימוש באנזימי רסטרקציה, אם כי ביעילות נמוכה[9]. ב-1997, חוקרים הצליחו לשפר את היעילות ורמת הדיוק של התהליך הקודם, באמצעות שימוש באנזים בשם Pfu DNA polymerase, אשר מגביר את רמת הדיוק של ריאקציית ה-PCR. הם השתמשו גם באנזים DpnI על מנת לעכל ("digestion") את המקטע שאינו מוטאנטי אחרי הליגציה (ראו בהמשך), ומהלך זה מנע יצירה של תוצרים חלקיים של תבנית הרצף ("Template") המקורית, והגביר את רמת היעילות. השיטה הייתה יעילה, אבל ריבוי השלבים הכלולים בה הגביל את מספר המוטציות שניתן להחדיר באמצעותה[10]. בשנת 2000, התפרסמה שיטה משופרת, תוך שימוש במערכת QuikChange. החוקרים השתמשו באנזים Pfu DNA polymerase עם פריימרים משלימים לשני גדילי ה-DNA. אחרי ריאקציית ה-PCR הם השתמשו באנזים DpnI כדי לבצע עיכול סלקטיבי ל-DNA שעבר מתילציה. כתוצאה מהמתילציה, רק ה-DNA בתבנית המקורית נחתך – זאת לעומת הרצף המוטאנטי שהקנה עמידות לעיכול הזה. היתרון בשיטה הזאת הוא האפשרות להכניס מוטציות מרובות בו זמנית. שיטה זו מהירה יותר, יעילה יותר ומדויקת יותר מקודמתה, והיא הפחיתה משמעותית את הזמן והמאמצים הנדרשים ליצירת מוטציות רבות בתבניות DNA ארוכות[10][11].
זו שיטה שפותחה לצורך הנדסה גנטית של גנים מקודדי חלבון עם אפשרות להחדיר מספר רב של מוטציות באתרים ספציפיים ב-DNA. בשיטה זו מכניסים מקטעי DNA המכילים את המוטציות הרצויות לתוך מקטע DNA גנומי. חוקרים השתמשו באנזימי רסטריקציה כדי לחתוך קרוב למקטעי המטרה של ה-DNA. תהליך זה גורם ליצירת חסר של רצף המטרה, שבמקומו מוחדר רצף DNA המכיל את המוטציות הרצויות עם קצוות משלימים לקצוות החיתוך התוחמים את שני צדדיו של הרצף שהוסר ברקומבינאציה הומולוגית. בסופו של דבר, המקטע המתקבל עובר ריצוף כדי לבדוק אם הוא מכיל את המוטציות שתוכננו[12][13]. במחקר שנעשה ב־1985, הצלחת השיטה הייתה מוגבלת, בשל יעילות נמוכה של הכנסת מוטציות חדשות לרצף נתון ברקומבינאציה הומולוגית, ובשל יצירת מוטציות נוספות לא רצויות[13]. בשנת 1988 הוכנסו מספר שיפורים לשיטה. החוקרים שיפרו את רמת הדיוק והיעילות על ידי שימוש באוליגונוקלאוטידים נקיים יותר ללא מלח, מה שהפחית את מספר המוטציות הלא רצויות ושיפר את רמת הדיוק. הם גם שינו את אתרי החיתוך המוכרים על ידי אנזימי רסטריקציה מדויקים יותר[14]. השיפורים הללו הפכו את התהליך למהיר, יעיל ומדויק יותר[15].
בהמשך הוכנסו עוד שיפורים לשיטת המוטגנזה באמצעות קסטת רצפים. בשנת 2020 החוקרים פיתחו את שיטת ה-RedEx: זו שיטה למוטגנזה מכוונת שמאפשרת הכנסה, מחיקה או החלפה של נוקלאוטידים, מקטעי DNA וגנים שלמים בתוך אתר-המטרה, ללא שינויים לא רצויים. השיטה כוללת הכנסה של קסטת RedEx המכילה מקטעים הומולוגיים משני צידי אתר המטרה, וכן של המוטציה הרצויה, גן עמידות לאמפיצילין וגן הרעלן ccdB לסלקציה, המאפשרים הכנסה מדויקת של שינויים בגנום באמצעות רקומבינציה הומולוגית. אחרי סלקציה לטובת תאים בעלי עמידות לאמפיצילין, מסירים את הסמנים באמצעות אנזימי רסטריקציה כדי לקבל DNA ליניארי. לאחר מכן נעשה שימוש באקסונוקלאז וליגציה (איחוי) כדי להפוך את ה-DNA הליניארי למעגלי. התהליך מסתיים בבדיקה אם המוטציה הרצויה נמצאת ברצף ה-DNA על ידי ביצוע PCR וריצוף[16][17]. השיפורים כוללים גם שימוש בקסטת RedEx עם רצפים הומולוגיים ייחודיים באורך 20 בסיסים הממוקמים בקצוות ה-DNA, מה שמונע רקומבינציה לא רצויה, ואף משפר את רמת הדיוק והיעילות של הכנסת המוטציות ללא השארת רצפים זרים[16].
שימושים במדע
עריכה- אבולוציה מבוקרת והנדסת חלבונים: שימוש במוטגנזה המכוונת למיקום ספציפי, כדי לייצר שינויים ברצפי ה-DNA, במטרה לחקור את תהליכי הברירה הטבעית וגם להאיץ את האבולוציה של חלבונים נבחרים ליצירת תכונות חדשות או לשיפור פעילותם. החוקרים מחדירים מוטציות ספציפיות לגנים מקודדי-חלבון, ולאחר מכן הם בוחנים את תכונות החלבונים המוטנטים בהשוואה לחלבון המקורי. ניסוי כזה מאפשר לברר את השינויים הדרושים לשיפור פעילות חלבון נחקר[18][19].
- הנדסת נוגדנים: המוטגנזה המכוונת למיקום מדויק מאפשרת לחוקרים לשפר את האפיניות והספציפיות של נוגדנים לאנטיגנים על ידי שינויים ממוקדים ומדויקים ברצף ה-DNA. בשיטת מוטגנזה מכוונת על ידי אוליגונוקלאוטידים ניתן גם להקטין את הרגישות. בנוסף, ניתן לייצר ספריות מנוהלות של נוגדנים, ולזהות וריאנטים בעלי אפיניות משופרת לאנטיגנים ספציפיים[20][21].
- מוטגנזה מכוונת בצמחים: שיטה זו מאפשרת יצירת שינויים ספציפיים ברצפי ה-DNA המקודדים לגנים נבחרים בצמחים, במטרה לשפר את התכונות הרצויות כמו עמידות למזיקים, או שיפור של תכונות תזונתיות ויכולת ההסתגלות לתנאי סביבה קשים. חוקרים יכולים לגרום למוטציות שמובילות לשינויים גנטיים תורשתיים בצמחים כמו תירס, חיטה ועגבנייה. התהליך כולל זיהוי רצף המטרה ב-DNA צמחי, וחיתוך ברצף ה-DNA בנקודות ספציפיות על ידי אנזימים. כך ניתן להכניס מוטציות שמעניקות לצמח תכונות משופרות, כמו עמידות למחלות והגדלה של היבול[22][23].
הערות שוליים
עריכה- ^ Michael P. Weiner, Gina L. Costa, Warren Schoettlin, Janice Cline, Eric Mathur, John C. Bauer, Site-directed mutagenesis of double-stranded DNA by the polymerase chain reaction, Gene 151, 1994-12-30, עמ' 119–123 doi: 10.1016/0378-1119(94)90641-6
- ^ 1 2 Michael P. Weiner, Gina L. Costa, Warren Schoettlin, Janice Cline, Eric Mathur, John C. Bauer, Site-directed mutagenesis of double-stranded DNA by the polymerase chain reaction, Gene 151, 1994-12, עמ' 119–123 doi: 10.1016/0378-1119(94)90641-6
- ^ Jian-Min Chen, David N. Cooper, Nadia Chuzhanova, Claude Férec, George P. Patrinos, Gene conversion: mechanisms, evolution and human disease, Nature Reviews Genetics 8, 2007-09-11, עמ' 762–775 doi: 10.1038/nrg2193
- ^ 1 2 3 C.A. Hutchison, S. Phillips, M.H. Edgell, S. Gillam, P. Jahnke, M. Smith, Mutagenesis at a specific position in a DNA sequence., Journal of Biological Chemistry 253, 1978-09, עמ' 6551–6560 doi: 10.1016/s0021-9258(19)46967-6
- ^ J W Taylor, J Ott, F Eckstein, The rapid generation of oligonucleotide-directed mutations at high frequency using phosphorothioate-modified DNA., Nucleic Acids Research 13, 1985-12-20, עמ' 8765–8785
- ^ T A Kunkel, Rapid and efficient site-specific mutagenesis without phenotypic selection., Proceedings of the National Academy of Sciences 82, 1985-01, עמ' 488–492 doi: 10.1073/pnas.82.2.488
- ^ Guoyi Zhang, Junyu Wang, Yiwen Li, Guangdong Shang, CRISPR/Cas9-assisted ssDNA recombineering for site-directed mutagenesis and saturation mutagenesis of plasmid-encoded genes, Biotechnology Letters 45, 2023-06-01, עמ' 629–637 doi: 10.1007/s10529-023-03363-1
- ^ Patrick D. Hsu, Eric S. Lander, Feng Zhang, Development and Applications of CRISPR-Cas9 for Genome Engineering, Cell 157, 2014-06-05, עמ' 1262–1278 doi: 10.1016/j.cell.2014.05.010
- ^ D Barettino, M Feigenbutz, R Valcárcel, H G Stunnenberg, Improved method for PCR-mediated site-directed mutagenesis., Nucleic Acids Research 22, 1994-02-11, עמ' 541–542
- ^ 1 2 Constance L. Fisher, Guo Kui Pei, Modification of a PCR-Based Site-Directed Mutagenesis Method, BioTechniques 23, 1997-10, עמ' 570–574 doi: 10.2144/97234bm01
- ^ Yang-Gyun Kim, Stefan Maas, Multiple Site Mutagenesis with High Targeting Efficiency in One Cloning Step, BioTechniques 28, 2000-02, עמ' 196–198 doi: 10.2144/00282bm01
- ^ G Dalbadie-McFarland, L W Cohen, A D Riggs, C Morin, K Itakura, J H Richards, Oligonucleotide-directed mutagenesis as a general and powerful method for studies of protein function., Proceedings of the National Academy of Sciences 79, 1982-11, עמ' 6409–6413 doi: 10.1073/pnas.79.21.6409
- ^ 1 2 James A. Wells, Mark Vasser, David B. Powers, Cassette mutagenesis: an efficient method for generation of multiple mutations at defined sites, Gene 34, 1985-01-01, עמ' 315–323 doi: 10.1016/0378-1119(85)90140-4
- ^ J. A. Wells, D. A. Estell, Subtilisin--an enzyme designed to be engineered, Trends in Biochemical Sciences 13, 1988-08, עמ' 291–297 doi: 10.1016/0968-0004(88)90121-1
- ^ Noel J. Sauer, Jerry Mozoruk, Ryan B. Miller, Zachary J. Warburg, Keith A. Walker, Peter R. Beetham, Christian R. Schöpke, Greg F. W. Gocal, Oligonucleotide‐directed mutagenesis for precision gene editing, Plant Biotechnology Journal 14, 2015-10-27, עמ' 496–502 doi: 10.1111/pbi.12496
- ^ 1 2 Ji Luan, Chaoyi Song, Yan Liu, Ruoting He, Ruofei Guo, Qingwen Cui, Chanjuan Jiang, Xiaochen Li, Kexin Hao, A. Francis Stewart, Jun Fu, Youming Zhang, Hailong Wang, Seamless site-directed mutagenesis in complex cloned DNA sequences using the RedEx method, Nature Protocols, 2024-07-15, עמ' 1–29 doi: 10.1038/s41596-024-01016-9
- ^ Chaoyi Song, Ji Luan, Ruijuan Li, Chanjuan Jiang, Yu Hou, Qingwen Cui, Tianqi Cui, Long Tan, Zaichao Ma, Ya-Jie Tang, A. Francis Stewart, Jun Fu, Youming Zhang, Hailong Wang, RedEx: a method for seamless DNA insertion and deletion in large multimodular polyketide synthase gene clusters, Nucleic Acids Research 48, 2020-12-16, עמ' e130 doi: 10.1093/nar/gkaa956
- ^ Eric A. Gaucher, Xun Gu, Michael M. Miyamoto, Steven A. Benner, Predicting functional divergence in protein evolution by site-specific rate shifts, Trends in Biochemical Sciences 27, 2002-06, עמ' 315–321 doi: 10.1016/s0968-0004(02)02094-7
- ^ Yuyan Bao, Yuanyuan Xu, Xiaoqiang Huang, Focused rational iterative site-specific mutagenesis (FRISM): A powerful method for enzyme engineering, Molecular Catalysis 553, 2024-01-15, עמ' 113755 doi: 10.1016/j.mcat.2023.113755
- ^ Mark L. Chiu, Dennis R. Goulet, Alexey Teplyakov, Gary L. Gilliland, Antibody Structure and Function: The Basis for Engineering Therapeutics, Antibodies 8, 2019-12, עמ' 55 doi: 10.3390/antib8040055
- ^ Robert F. Balint, James W. Larrick, Antibody engineering by parsimonious mutagenesis, Gene 137, 1993-12-27, עמ' 109–118 doi: 10.1016/0378-1119(93)90258-5
- ^ Si Nian Char, Erica Unger‐Wallace, Bronwyn Frame, Sarah A. Briggs, Marcy Main, Martin H. Spalding, Erik Vollbrecht, Kan Wang, Bing Yang, Heritable site‐specific mutagenesis using TALEN s in maize, Plant Biotechnology Journal 13, 2015-09, עמ' 1002–1010 doi: 10.1111/pbi.12344
- ^ Yusuff Oladosu, Mohd Y. Rafii, Norhani Abdullah, Ghazali Hussin, Asfaliza Ramli, Harun A. Rahim, Gous Miah, Magaji Usman, Principle and application of plant mutagenesis in crop improvement: a review, Biotechnology & Biotechnological Equipment 30, 2016-01-02, עמ' 1–16 doi: 10.1080/13102818.2015.1087333